# Importation des packages
import pandas as pd
import numpy as np
import pandas as pd
import chardet
import warnings
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
warnings.filterwarnings("ignore")
from sklearn.model_selection import train_test_split
from sklearn.feature_selection import mutual_info_classif
from sklearn.feature_selection import mutual_info_classif
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler, OneHotEncoder
from sklearn.impute import SimpleImputer
from sklearn.compose import ColumnTransformer
from sklearn.pipeline import Pipeline
from sklearn.linear_model import LogisticRegression, LogisticRegressionCV
from sklearn.metrics import ConfusionMatrixDisplay
from sklearn.metrics import accuracy_score
from sklearn.metrics import confusion_matrix
from sklearn.ensemble import RandomForestClassifier
from sklearn.preprocessing import LabelEncoder
import xgboost as xgb
from xgboost import XGBClassifierExamen de machine learning
Introduction
Il est possible que ce soit Da Veiga qui assure vos cours, mais je vais vous offrir un aperçu de ce à quoi pourrait ressembler votre examen. C’est important, car nous avons tendance à sous-estimer ce type d’activité, surtout lorsqu’il autorise l’utilisation de générateurs de texte tels que ChatGPT. Cette année, peu d’étudiants ont achevé le projet, la charge de données étant longue et fastidieuse. Je vous conseille de vous y prendre en avance. Préparez des fonctions exécutant certaines tâches spécifiques, que je vous expliquerai progressivement.
Les données
Les données, très larges proposées sont Dataset1 et Dataset2. Vous les trouverez sur mon github
Packages
J’utiliserai les packages suivants: sklearn, pandas, numpy, matplotlib,seaborn pour la visualiation.
Objectif
L’objectif de ce notebook consistera à une ’analyse exploratoire des données, le prétraitement des données pour la modélisation, et l’application ainsi que l’évaluation de modèles de classification() sur un ensemble de données.
Chargement des données
with open('Dataset1.csv', 'rb') as f:
result = chardet.detect(f.read()) # or readline if the file is large
#print(result['encoding'])
Dataset1 = pd.read_csv('Dataset1.csv', delimiter=",",decimal = ".",encoding=result['encoding'])
Dataset1.head()| Vegetation_Type_2 | Groundwater_Level_1 | Drainage_Quality_1 | Slope | Hillshade_9am | Hillshade_Noon | Pollution_Level_1 | Water_Source_Distance_2 | Terrain_Roughness_2 | Urban_Proximity_Index_1 | ... | Soil_Moisture_Level_2 | Horizontal_Distance_To_Hydrology | Wind_Speed_Average_2 | Elevation_Range_1 | Pollution_Level_2 | Vegetation_Type_1 | Temperature_Average_1 | Canopy_Cover_2 | Elevation | Cover_Type | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 450.684968 | 2054.426315 | 229.994980 | 3 | 221 | 232 | 181.000000 | 227.161401 | 216.000000 | 14.0 | ... | 418.375833 | 258 | 116.000000 | 219.000000 | 224.000000 | 0.000000 | 9.000000 | 503.586301 | 2596 | 5 |
| 1 | 889.885392 | 997.573983 | 208.000000 | 2 | 220 | 235 | 166.000000 | 933.851870 | 251.000000 | 16.0 | ... | 2488.726342 | 212 | 179.025367 | 236.000000 | 224.000000 | 69.000000 | 17.000000 | 537.000000 | 2590 | 5 |
| 2 | 1232.738700 | 676.352745 | 214.313227 | 9 | 234 | 238 | 205.000000 | 4235.282432 | 176.898683 | 13.0 | ... | 1852.423116 | 268 | 156.000000 | 200.872578 | 230.415218 | 120.357544 | 10.000000 | 376.000000 | 2804 | 2 |
| 3 | 3359.512595 | 4720.481538 | 230.000000 | 18 | 238 | 238 | 176.002696 | 1066.935784 | 220.000000 | 11.0 | ... | 5388.528248 | 242 | 156.000000 | 230.000000 | 243.000000 | 15.000000 | 12.933234 | 30.000000 | 2785 | 2 |
| 4 | 1907.275049 | 2187.627994 | 221.000000 | 2 | 220 | 234 | 109.000000 | 1182.489702 | 184.859965 | 9.0 | ... | 304.080537 | 153 | 112.000000 | 232.000000 | 213.000000 | 39.000000 | 14.000000 | 330.000000 | 2595 | 5 |
5 rows × 51 columns
#info
Dataset1.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 581012 entries, 0 to 581011
Data columns (total 51 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Vegetation_Type_2 581012 non-null float64
1 Groundwater_Level_1 581012 non-null float64
2 Drainage_Quality_1 581012 non-null float64
3 Slope 581012 non-null int64
4 Hillshade_9am 581012 non-null int64
5 Hillshade_Noon 581012 non-null int64
6 Pollution_Level_1 581012 non-null float64
7 Water_Source_Distance_2 581012 non-null float64
8 Terrain_Roughness_2 581012 non-null float64
9 Urban_Proximity_Index_1 581012 non-null float64
10 Sunlight_Intensity_2 581012 non-null float64
11 Rainfall_Index_1 581012 non-null float64
12 Canopy_Cover_1 581012 non-null float64
13 Rock_Type_2 581012 non-null float64
14 Rainfall_Index_2 581012 non-null float64
15 Terrain_Slope_Angle_2 581012 non-null float64
16 Temperature_Average_2 581012 non-null float64
17 Air_Quality_Index_1 581012 non-null float64
18 Horizontal_Distance_To_Roadways 581012 non-null int64
19 Soil_Mineral_Content_2 581012 non-null float64
20 Wildlife_Density_2 581012 non-null float64
21 Water_Source_Distance_1 581012 non-null float64
22 Air_Quality_Index_2 581012 non-null float64
23 Wildlife_Density_1 581012 non-null float64
24 Drainage_Quality_2 581012 non-null float64
25 Land_Use_Category_2 581012 non-null float64
26 Groundwater_Level_2 581012 non-null float64
27 Wind_Speed_Average_1 581012 non-null float64
28 Hillshade_3pm 581012 non-null int64
29 Terrain_Slope_Angle_1 581012 non-null float64
30 Land_Use_Category_1 581012 non-null float64
31 Elevation_Range_2 581012 non-null float64
32 Vertical_Distance_To_Hydrology 581012 non-null int64
33 Urban_Proximity_Index_2 581012 non-null float64
34 Horizontal_Distance_To_Fire_Points 581012 non-null int64
35 Soil_Mineral_Content_1 581012 non-null float64
36 Aspect 581012 non-null int64
37 Soil_Moisture_Level_1 581012 non-null float64
38 Terrain_Roughness_1 581012 non-null float64
39 Rock_Type_1 581012 non-null float64
40 Sunlight_Intensity_1 581012 non-null float64
41 Soil_Moisture_Level_2 581012 non-null float64
42 Horizontal_Distance_To_Hydrology 581012 non-null int64
43 Wind_Speed_Average_2 581012 non-null float64
44 Elevation_Range_1 581012 non-null float64
45 Pollution_Level_2 581012 non-null float64
46 Vegetation_Type_1 581012 non-null float64
47 Temperature_Average_1 581012 non-null float64
48 Canopy_Cover_2 581012 non-null float64
49 Elevation 581012 non-null int64
50 Cover_Type 581012 non-null int64
dtypes: float64(40), int64(11)
memory usage: 226.1 MBLes données contiennent 51 colonnes et 581012 lignes. Dans le code ci-dessous, nous allons séparer les données en output(Y) et en features(X). L’output ici est la variable Cover_Type.
# split data into training and testing sets
X = Dataset1.drop(['Cover_Type'], axis=1).copy()
Y = Dataset1['Cover_Type'].copy()La prémiere chose à faire est de voir la distribution de Y. Afin de vérifier si c’est une variable catégorielle ou continue.
# Distribution de Y
sns.countplot(x='Cover_Type', data=Dataset1)
plt.show()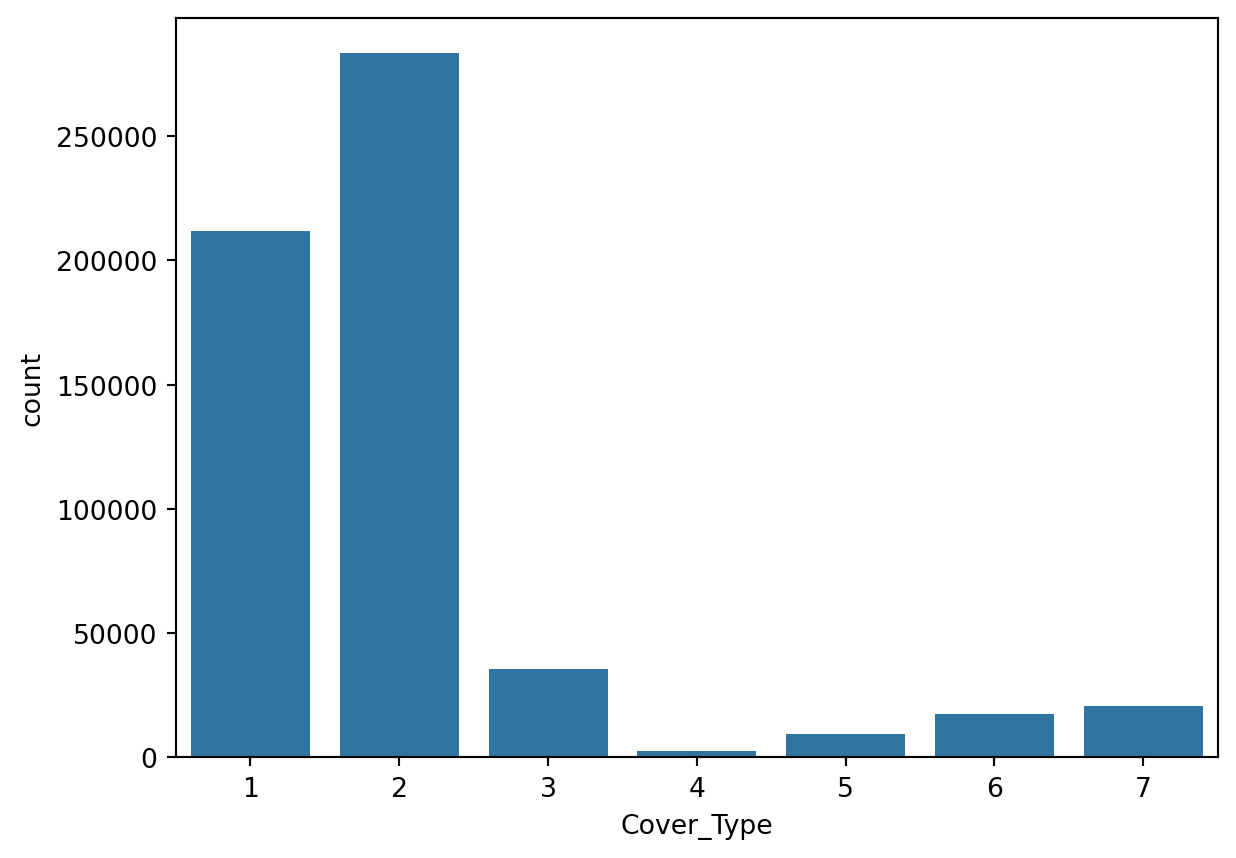
On peut voir que la variable est catégorielle. Elle contient 7 classes non équilibrées. On peut s’attendre que le modèle prédira mieux les classes les plus représentées(1 et 2) que les autres.
Nous ne traiterons pas ça ici. Les données étant labélisées, nous allons utiliser des modèles de classification.
Data preprocessing
Nous pouvons dans un premier temps vérifier s’il y a des valeurs manquantes dans les données.
# Missing values
Dataset1.isnull().sum()Vegetation_Type_2 0
Groundwater_Level_1 0
Drainage_Quality_1 0
Slope 0
Hillshade_9am 0
Hillshade_Noon 0
Pollution_Level_1 0
Water_Source_Distance_2 0
Terrain_Roughness_2 0
Urban_Proximity_Index_1 0
Sunlight_Intensity_2 0
Rainfall_Index_1 0
Canopy_Cover_1 0
Rock_Type_2 0
Rainfall_Index_2 0
Terrain_Slope_Angle_2 0
Temperature_Average_2 0
Air_Quality_Index_1 0
Horizontal_Distance_To_Roadways 0
Soil_Mineral_Content_2 0
Wildlife_Density_2 0
Water_Source_Distance_1 0
Air_Quality_Index_2 0
Wildlife_Density_1 0
Drainage_Quality_2 0
Land_Use_Category_2 0
Groundwater_Level_2 0
Wind_Speed_Average_1 0
Hillshade_3pm 0
Terrain_Slope_Angle_1 0
Land_Use_Category_1 0
Elevation_Range_2 0
Vertical_Distance_To_Hydrology 0
Urban_Proximity_Index_2 0
Horizontal_Distance_To_Fire_Points 0
Soil_Mineral_Content_1 0
Aspect 0
Soil_Moisture_Level_1 0
Terrain_Roughness_1 0
Rock_Type_1 0
Sunlight_Intensity_1 0
Soil_Moisture_Level_2 0
Horizontal_Distance_To_Hydrology 0
Wind_Speed_Average_2 0
Elevation_Range_1 0
Pollution_Level_2 0
Vegetation_Type_1 0
Temperature_Average_1 0
Canopy_Cover_2 0
Elevation 0
Cover_Type 0
dtype: int64Il n’y a pas de valeurs manquantes dans les données.
Features selections.
Nous allons supprimer les variables en se basant sur le critère de l’information mutulle qui n’apportent pas au moins 0.01 d’information à la variable cible.
En termes simples, l’information mutuelle mesure combien la connaissance d’une variable réduit l’incertitude concernant l’autre.
mutual_info = mutual_info_classif(X, Y)
mutual_info_df = pd.DataFrame(mutual_info, index=X.columns, columns=['Mutual Information'])
# Filtrer les caractéristiques avec une information mutuelle supérieure à 0.01
relevant_features = mutual_info_df[mutual_info_df['Mutual Information'] > 0.01].index
# Afficher les caractéristiques pertinentes
print("Caractéristiques pertinentes (Information Mutuelle > 0.01) :")
print(relevant_features)Caractéristiques pertinentes (Information Mutuelle > 0.01) :
Index(['Slope', 'Hillshade_9am', 'Hillshade_Noon',
'Horizontal_Distance_To_Roadways', 'Hillshade_3pm',
'Vertical_Distance_To_Hydrology', 'Horizontal_Distance_To_Fire_Points',
'Aspect', 'Horizontal_Distance_To_Hydrology', 'Elevation'],
dtype='object')Nous allons supprimer les variables qui ne sont pas pertinentes.
# Supprimer les caractéristiques non pertinentes
X = X[relevant_features]
X.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 581012 entries, 0 to 581011
Data columns (total 10 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Slope 581012 non-null int64
1 Hillshade_9am 581012 non-null int64
2 Hillshade_Noon 581012 non-null int64
3 Horizontal_Distance_To_Roadways 581012 non-null int64
4 Hillshade_3pm 581012 non-null int64
5 Vertical_Distance_To_Hydrology 581012 non-null int64
6 Horizontal_Distance_To_Fire_Points 581012 non-null int64
7 Aspect 581012 non-null int64
8 Horizontal_Distance_To_Hydrology 581012 non-null int64
9 Elevation 581012 non-null int64
dtypes: int64(10)
memory usage: 44.3 MBLà on a plus que 10 features et les données sont toutes numériques.
X.describe()| Slope | Hillshade_9am | Hillshade_Noon | Horizontal_Distance_To_Roadways | Hillshade_3pm | Vertical_Distance_To_Hydrology | Horizontal_Distance_To_Fire_Points | Aspect | Horizontal_Distance_To_Hydrology | Elevation | |
|---|---|---|---|---|---|---|---|---|---|---|
| count | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 | 581012.000000 |
| mean | 14.103704 | 212.146049 | 223.318716 | 2350.146611 | 142.528263 | 46.418855 | 1980.291226 | 155.656807 | 269.428217 | 2959.365301 |
| std | 7.488242 | 26.769889 | 19.768697 | 1559.254870 | 38.274529 | 58.295232 | 1324.195210 | 111.913721 | 212.549356 | 279.984734 |
| min | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | -173.000000 | 0.000000 | 0.000000 | 0.000000 | 1859.000000 |
| 25% | 9.000000 | 198.000000 | 213.000000 | 1106.000000 | 119.000000 | 7.000000 | 1024.000000 | 58.000000 | 108.000000 | 2809.000000 |
| 50% | 13.000000 | 218.000000 | 226.000000 | 1997.000000 | 143.000000 | 30.000000 | 1710.000000 | 127.000000 | 218.000000 | 2996.000000 |
| 75% | 18.000000 | 231.000000 | 237.000000 | 3328.000000 | 168.000000 | 69.000000 | 2550.000000 | 260.000000 | 384.000000 | 3163.000000 |
| max | 66.000000 | 254.000000 | 254.000000 | 7117.000000 | 254.000000 | 601.000000 | 7173.000000 | 360.000000 | 1397.000000 | 3858.000000 |
analyse exploratoire des données
Pour l’analyse exploratoire, nous allons utiliser qu’un échantillon des données. Je vais en prendre 500
sample_size = 500 # Adjust this based on your dataset size
X_sampled = X.sample(n=sample_size, random_state=42)
Y_sampled = Y.loc[X_sampled.index]
X_sampled.describe()| Slope | Hillshade_9am | Hillshade_Noon | Horizontal_Distance_To_Roadways | Hillshade_3pm | Vertical_Distance_To_Hydrology | Horizontal_Distance_To_Fire_Points | Aspect | Horizontal_Distance_To_Hydrology | Elevation | |
|---|---|---|---|---|---|---|---|---|---|---|
| count | 500.000000 | 500.000000 | 500.000000 | 500.000000 | 500.000000 | 500.000000 | 500.00000 | 500.000000 | 500.000000 | 500.000000 |
| mean | 14.642000 | 211.054000 | 223.252000 | 2313.700000 | 143.338000 | 46.152000 | 1906.97800 | 155.922000 | 271.758000 | 2945.782000 |
| std | 7.678265 | 28.780405 | 19.804366 | 1548.527391 | 40.718494 | 60.782744 | 1297.58581 | 111.414732 | 227.100543 | 290.643937 |
| min | 1.000000 | 75.000000 | 149.000000 | 85.000000 | 0.000000 | -152.000000 | 42.00000 | 0.000000 | 0.000000 | 1983.000000 |
| 25% | 9.000000 | 197.000000 | 212.750000 | 1086.250000 | 119.000000 | 8.000000 | 982.75000 | 59.000000 | 108.000000 | 2805.000000 |
| 50% | 13.000000 | 217.000000 | 226.000000 | 1945.500000 | 142.000000 | 29.000000 | 1642.50000 | 125.000000 | 212.000000 | 3001.000000 |
| 75% | 19.000000 | 232.000000 | 237.000000 | 3226.250000 | 171.000000 | 67.000000 | 2411.25000 | 257.000000 | 376.750000 | 3149.250000 |
| max | 42.000000 | 253.000000 | 254.000000 | 7078.000000 | 246.000000 | 387.000000 | 6576.00000 | 359.000000 | 1110.000000 | 3529.000000 |
Nous pouvons tracer la distribution de chaque variable.
_ = X_sampled.hist(figsize=(20, 14))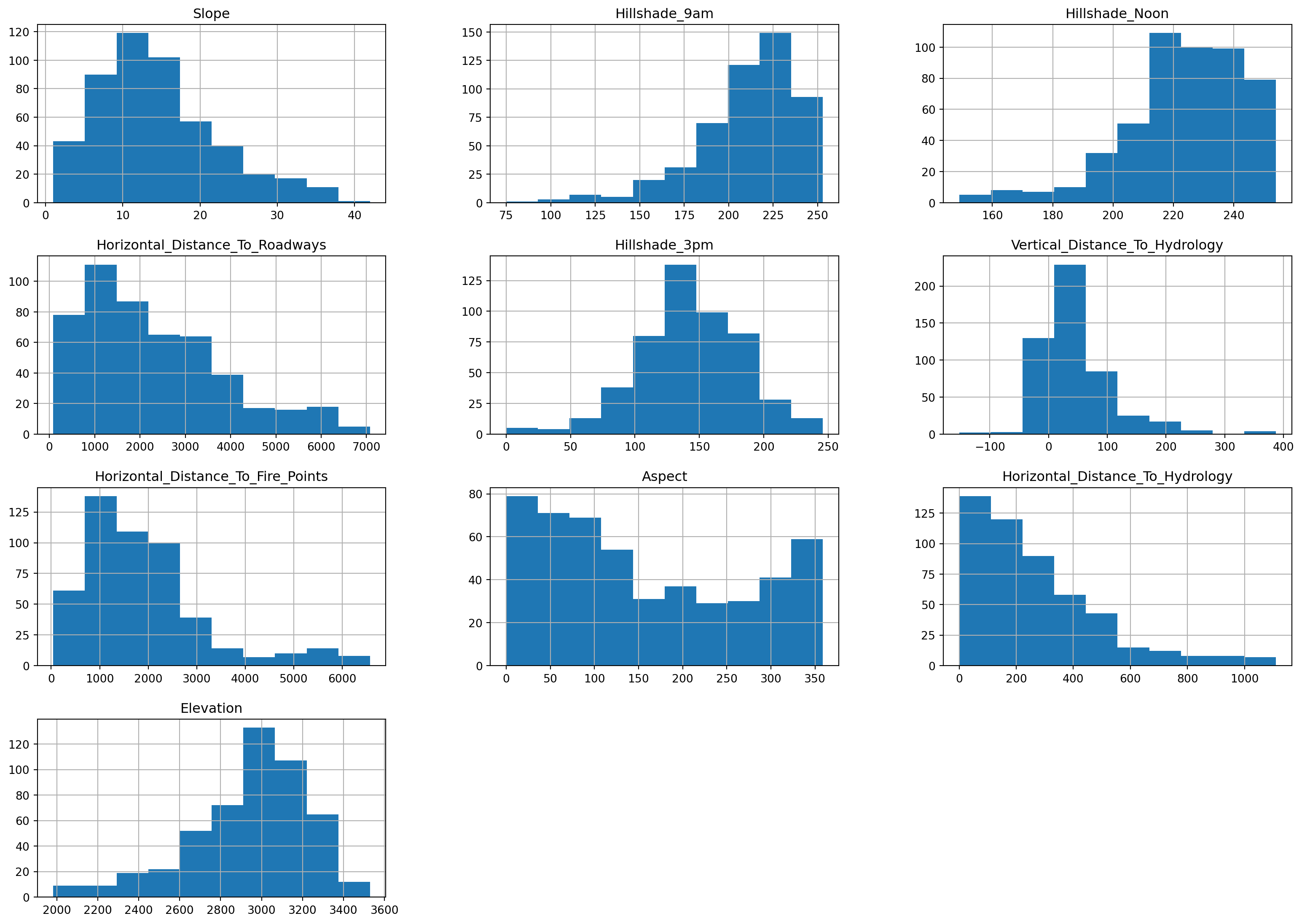
Voyons si on des dinausores dans les données.
sns.pairplot(X_sampled)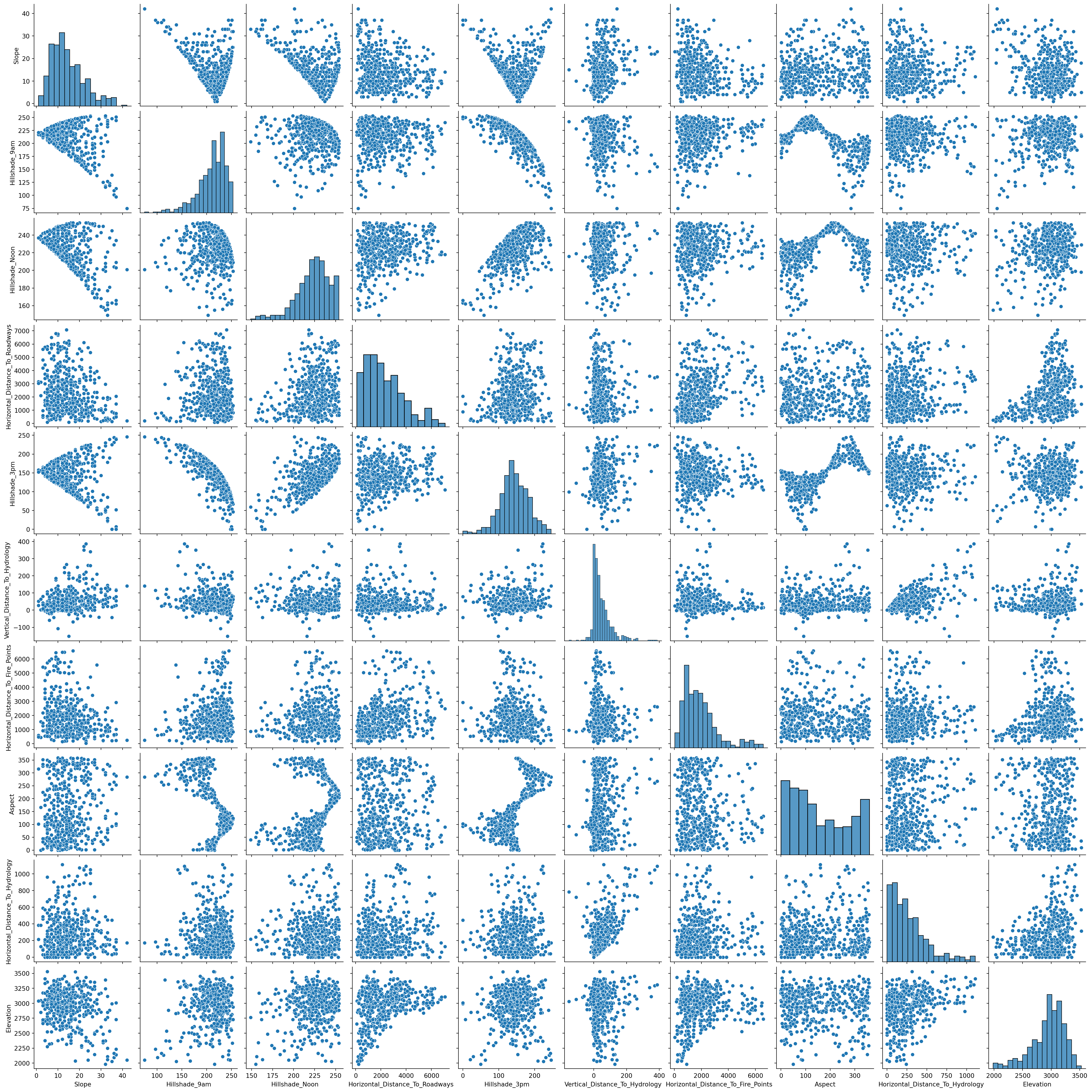
Il semble qu’il n’existe pas de corrélations linéaires entre les variables.
# Correlation matrix
import seaborn as sns
corr_matrix = X_sampled.corr()
# Heatmap of the correlation matrix
_= sns.heatmap(corr_matrix, annot=True)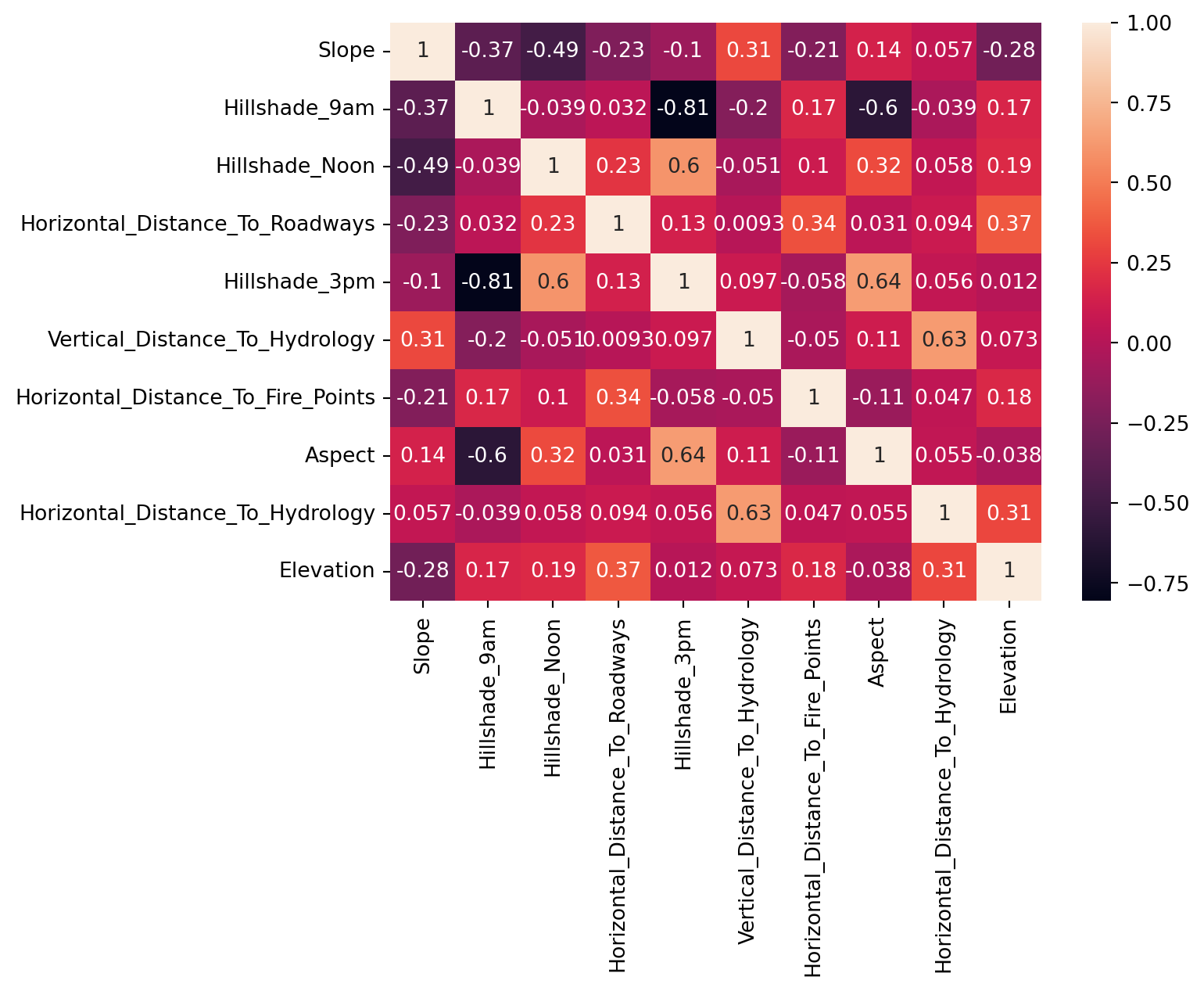
Ce graphique semble confirmer qu’il n’y a pas de corrélations linéaires entre les variables.
Box plot
Le graphique ci-dessous montre la distribution de chaque variable en fonction de la variable cible.
# box plot
# Transformed Cover_Type to categorical
Y_sampled = Y_sampled.astype('category')
for col in X_sampled.columns:
sns.boxplot(x=Y_sampled, y=X_sampled[col])
plt.show()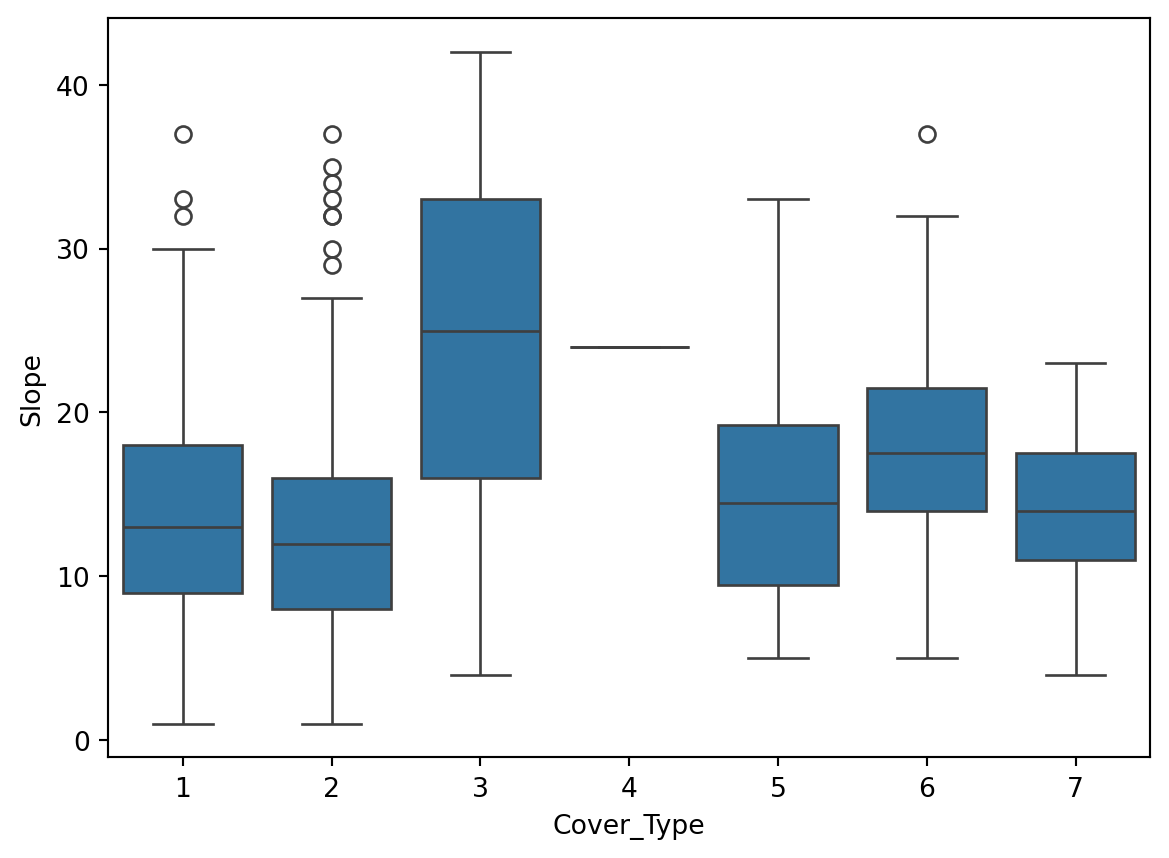
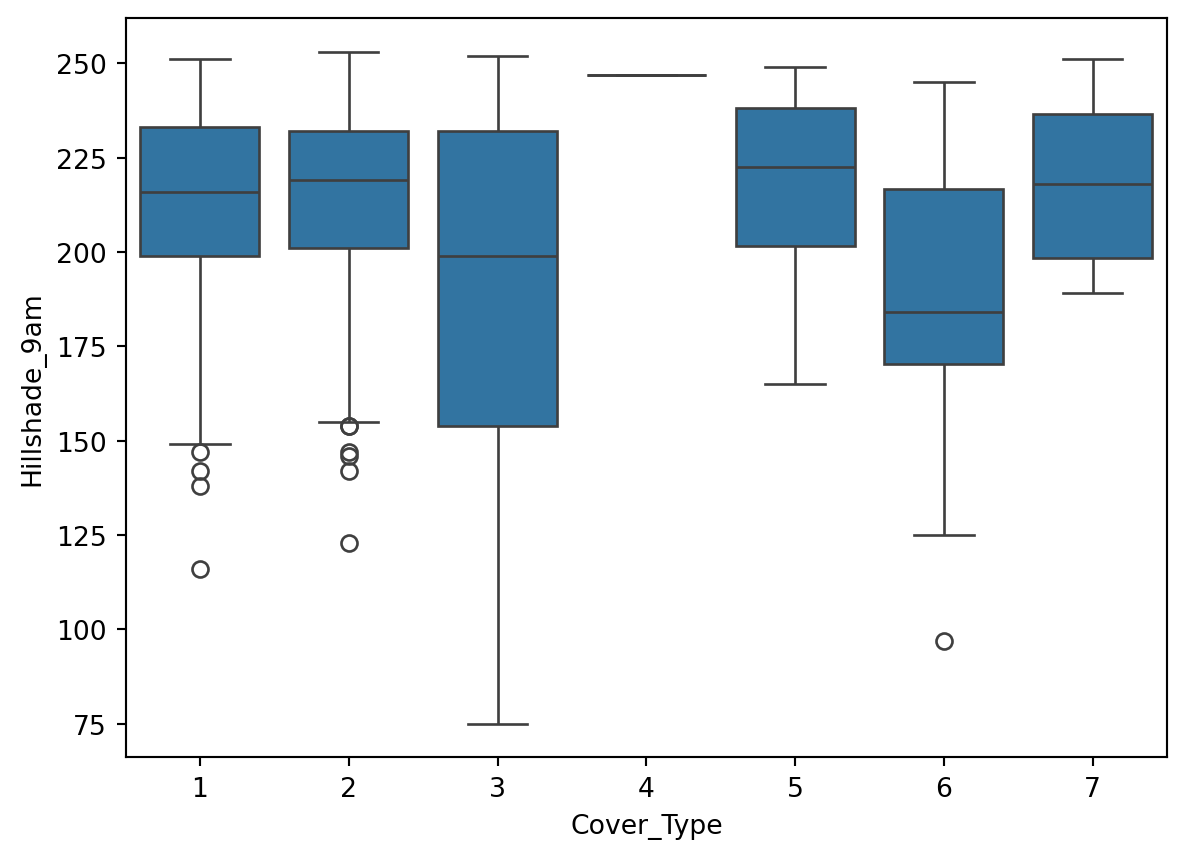
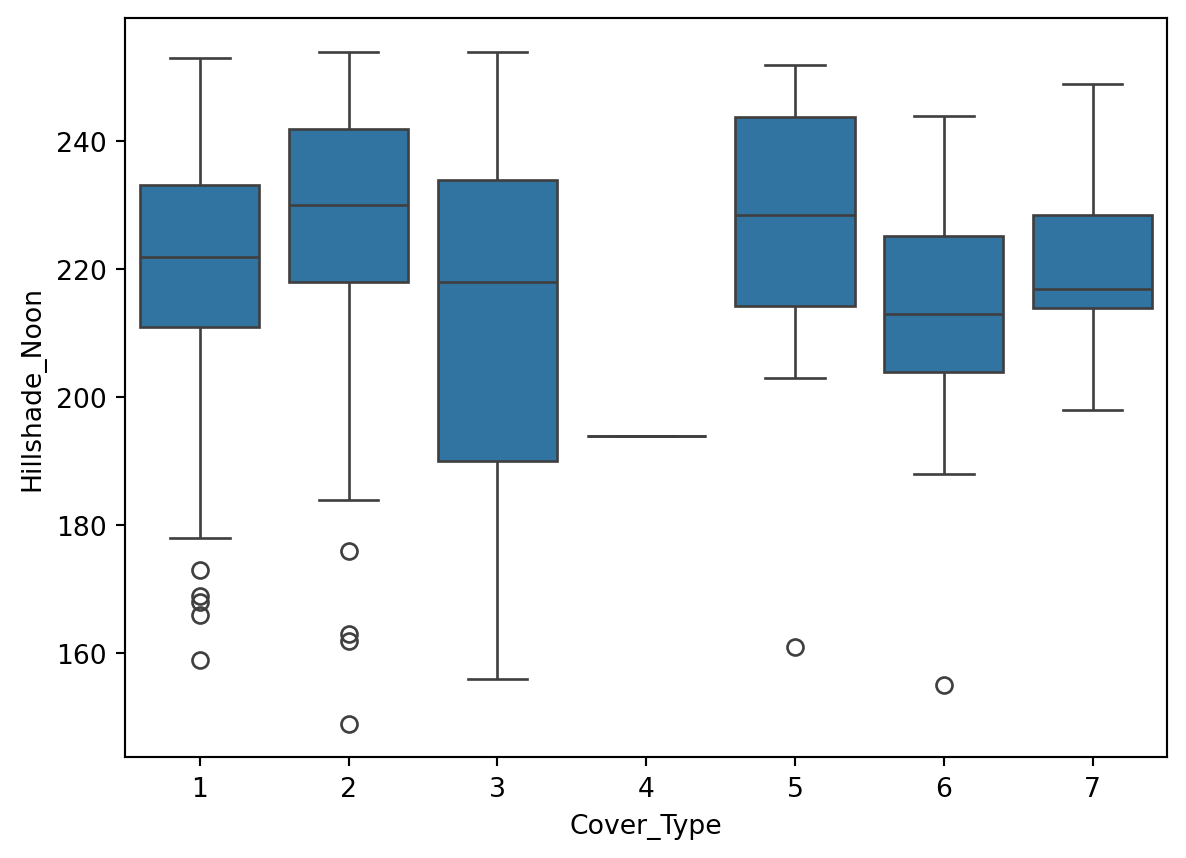
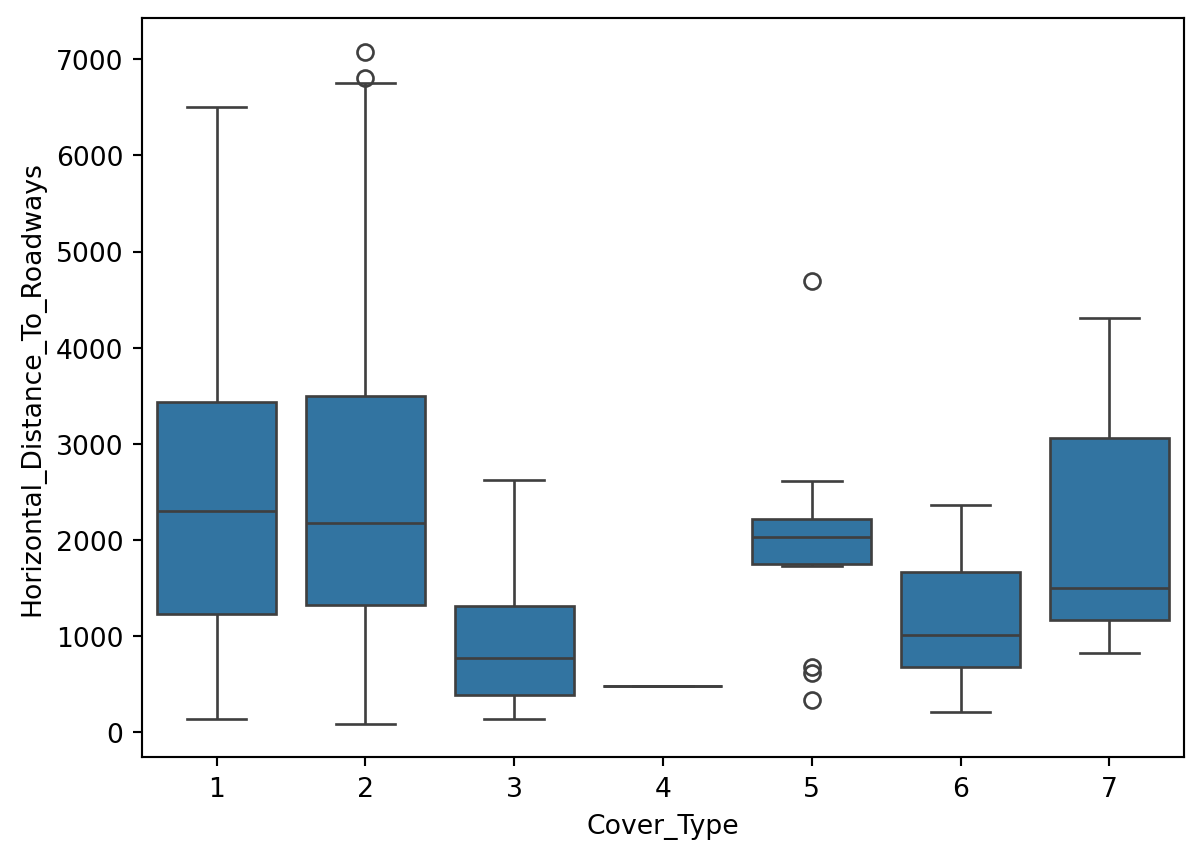
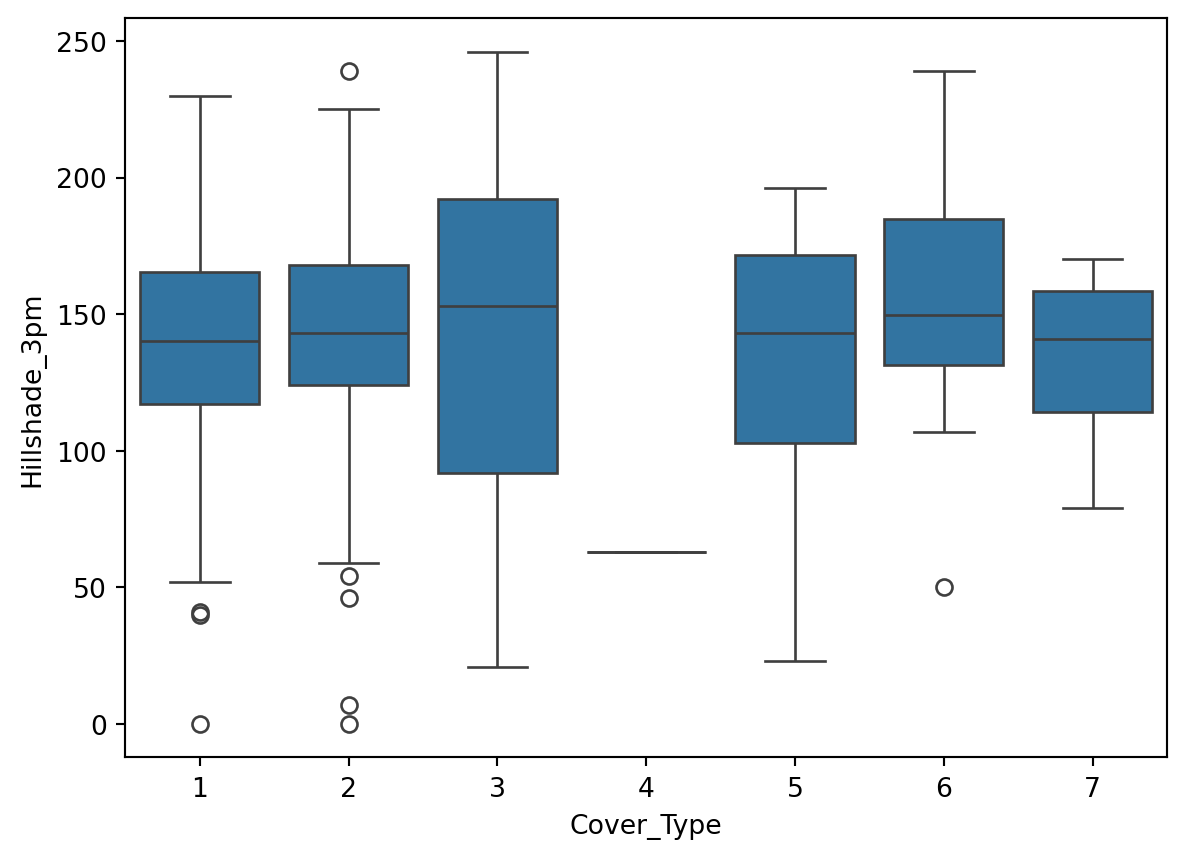
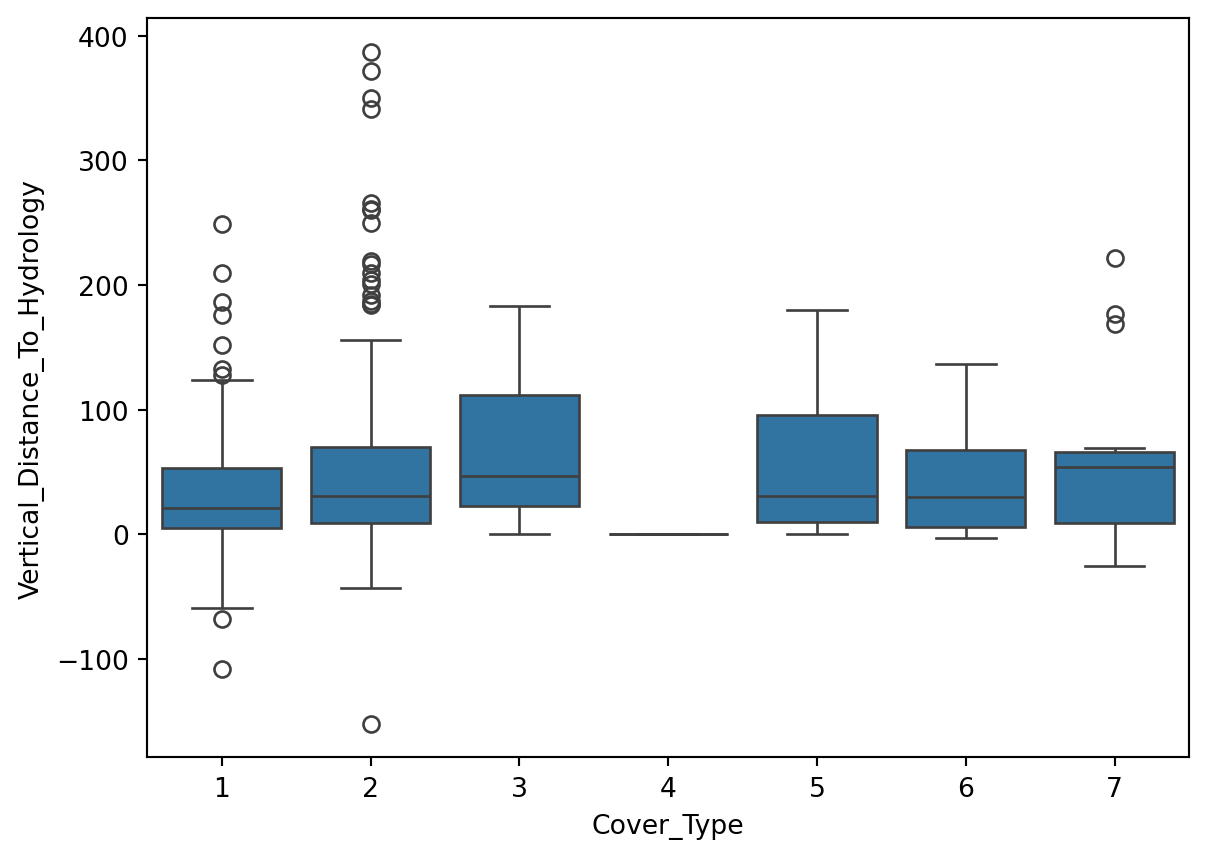
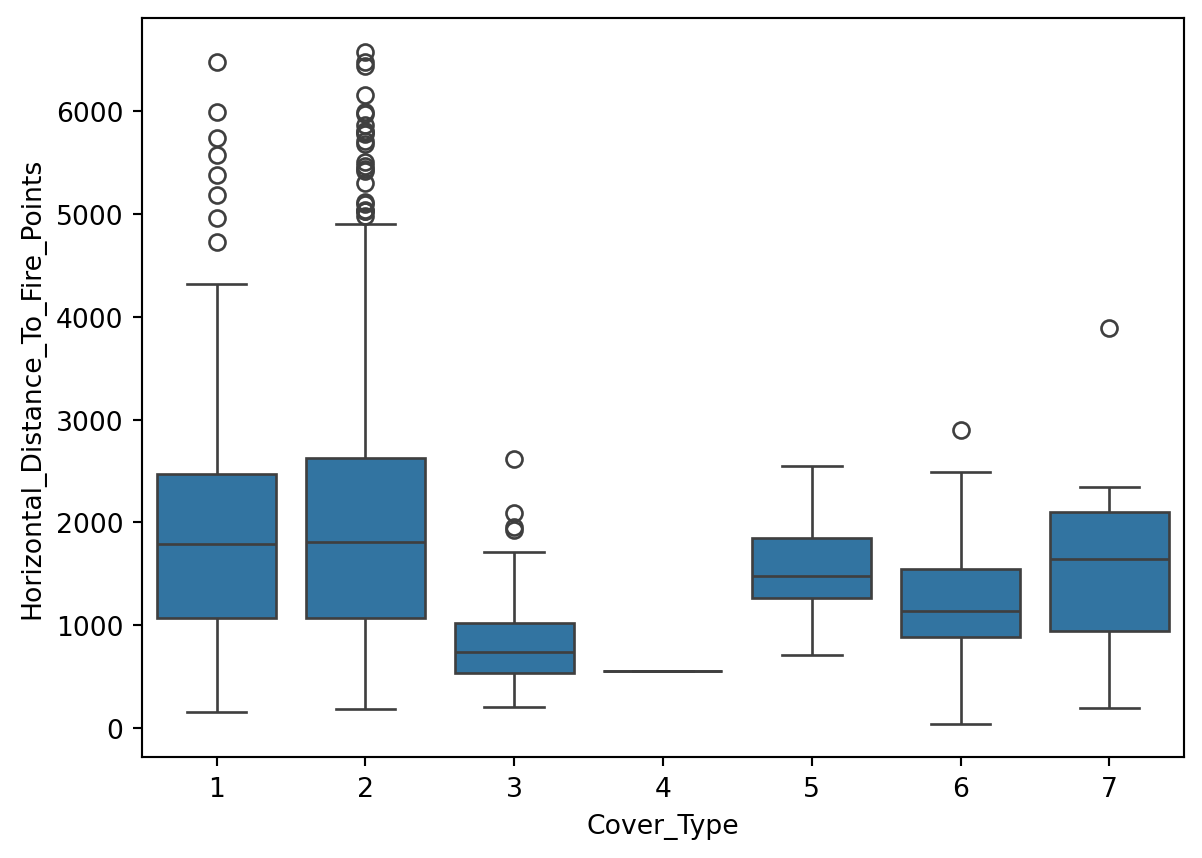
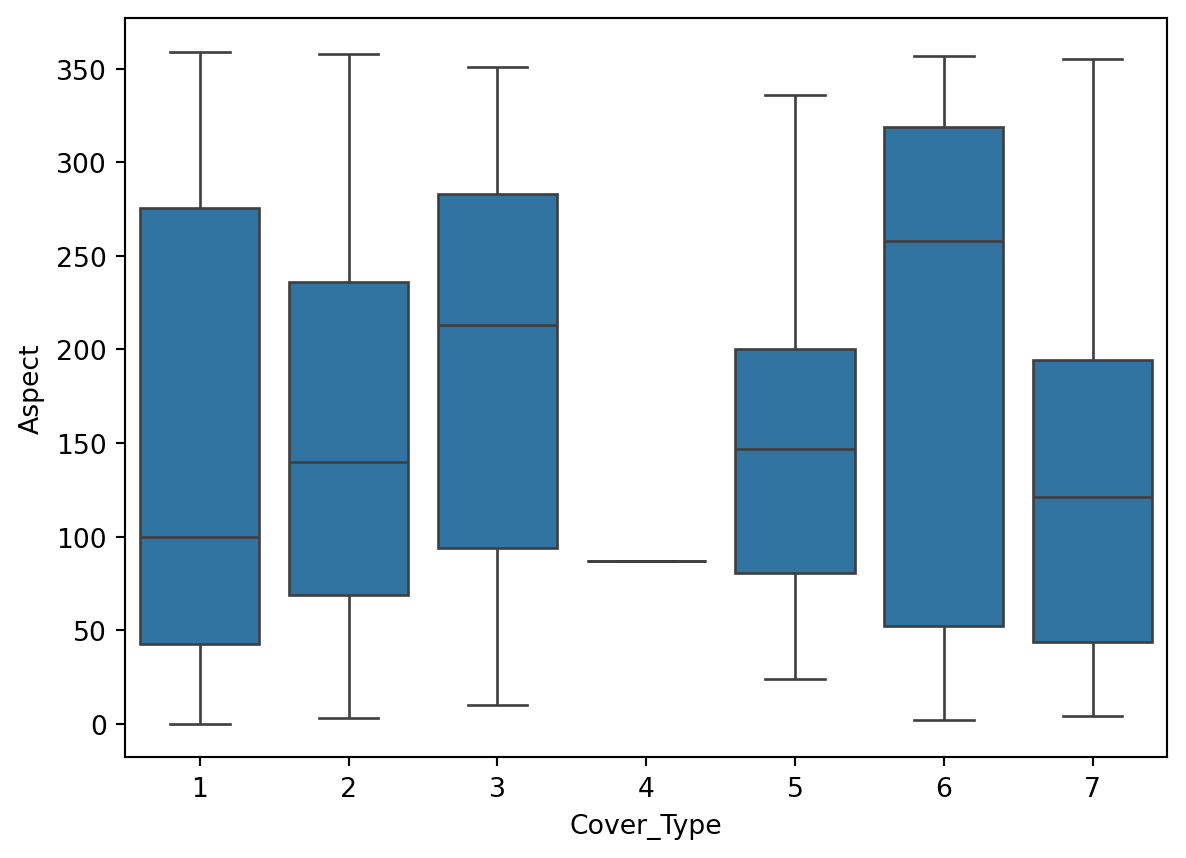
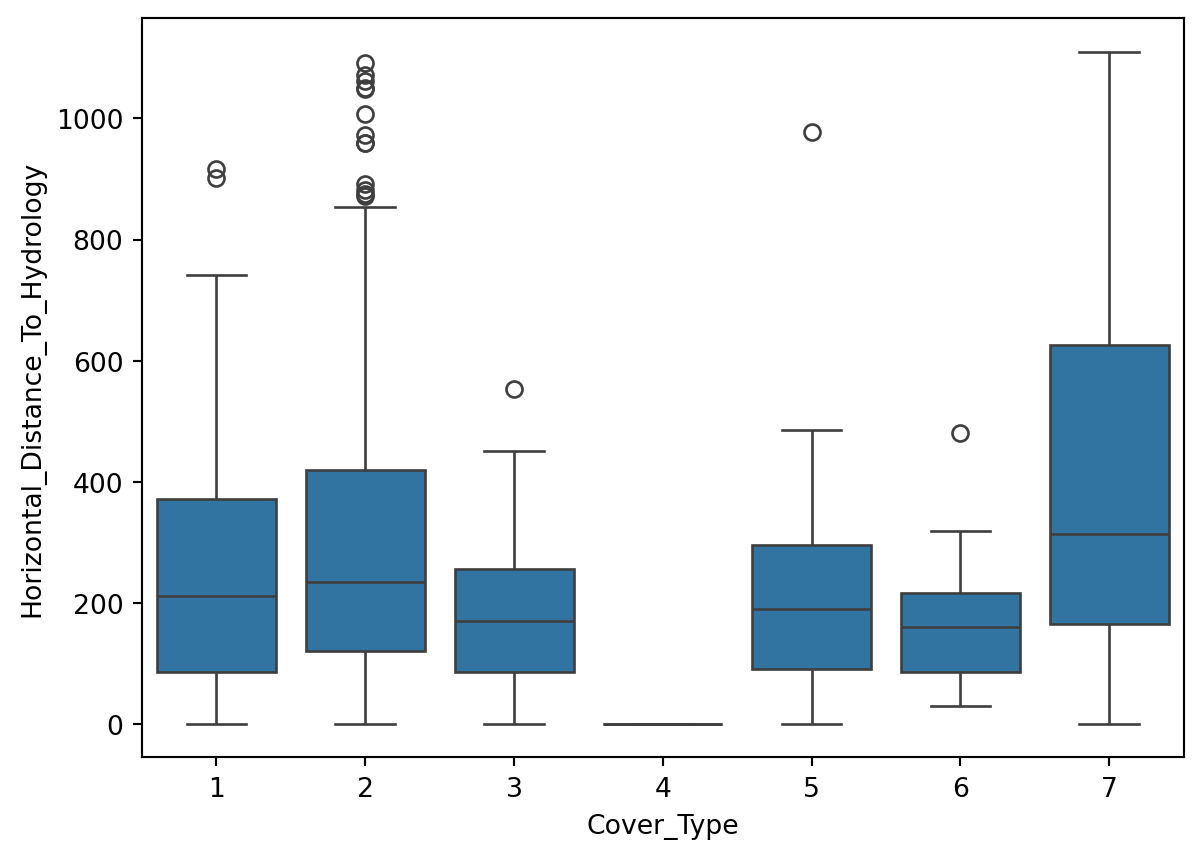
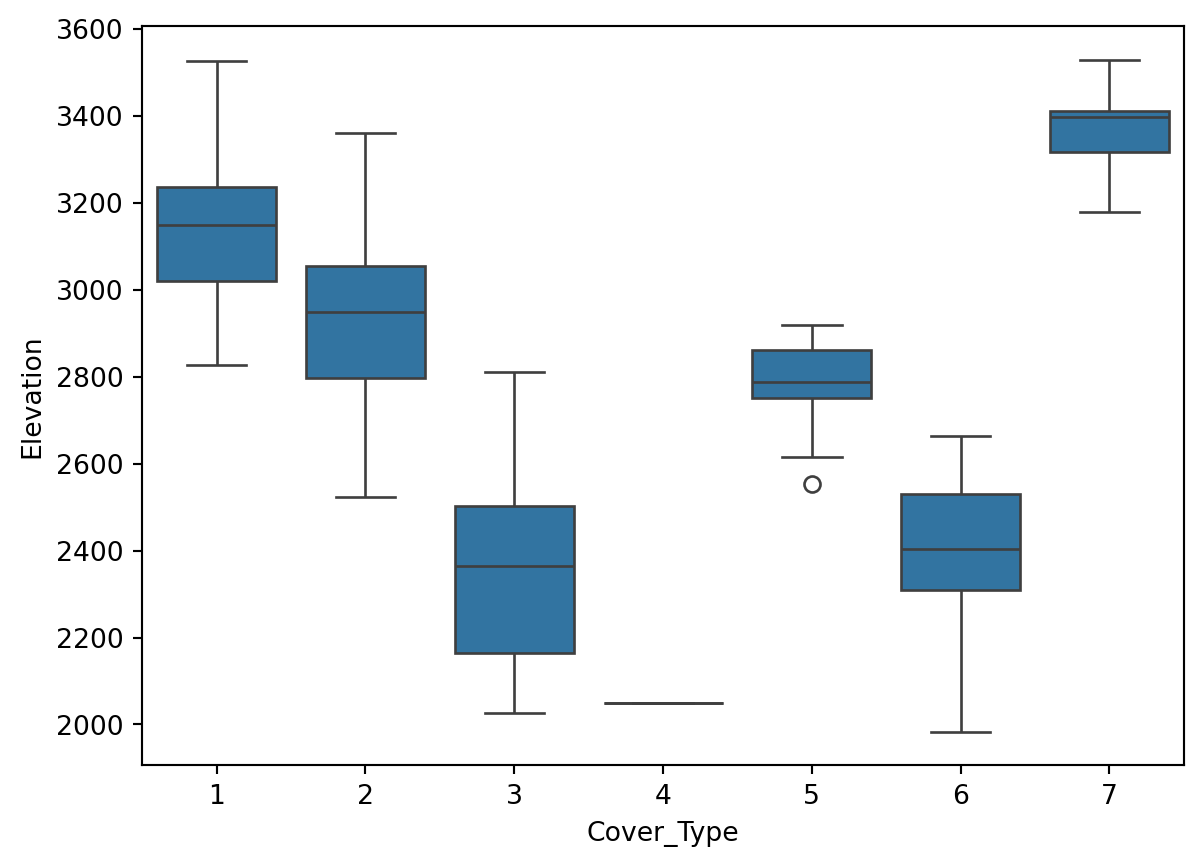
Interprétation: Nous allons nous concentrer sur la variable Elevation.
On peut voir que la variable Elevation est très discriminante.
Les types de couverture 1, 2, et 7 montrent des médianes relativement élevées pour l’élévation, avec 7 ayant la médiane la plus élevée, suivie par 1 et 2.
Chargement des données
Dataset2.
with open('Dataset2.csv', 'rb') as f:
result = chardet.detect(f.read()) # or readline if the file is large
#print(result['encoding'])
Dataset2 = pd.read_csv('Dataset2.csv', delimiter=",",decimal = ".",encoding=result['encoding'])
Dataset2.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 581012 entries, 0 to 581011
Data columns (total 2 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Wilderness_Area 581012 non-null int64
1 Soil_Type 581012 non-null int64
dtypes: int64(2)
memory usage: 8.9 MBCette fois-ci, nous avons 2 variables et 581012 lignes. On peut voir que les données sont de types int64 Inspectons ces données les pour voir si elles sont numériques ou catégorielles.
# Distribution de Y
sns.countplot(x='Wilderness_Area', data=Dataset2)
plt.show()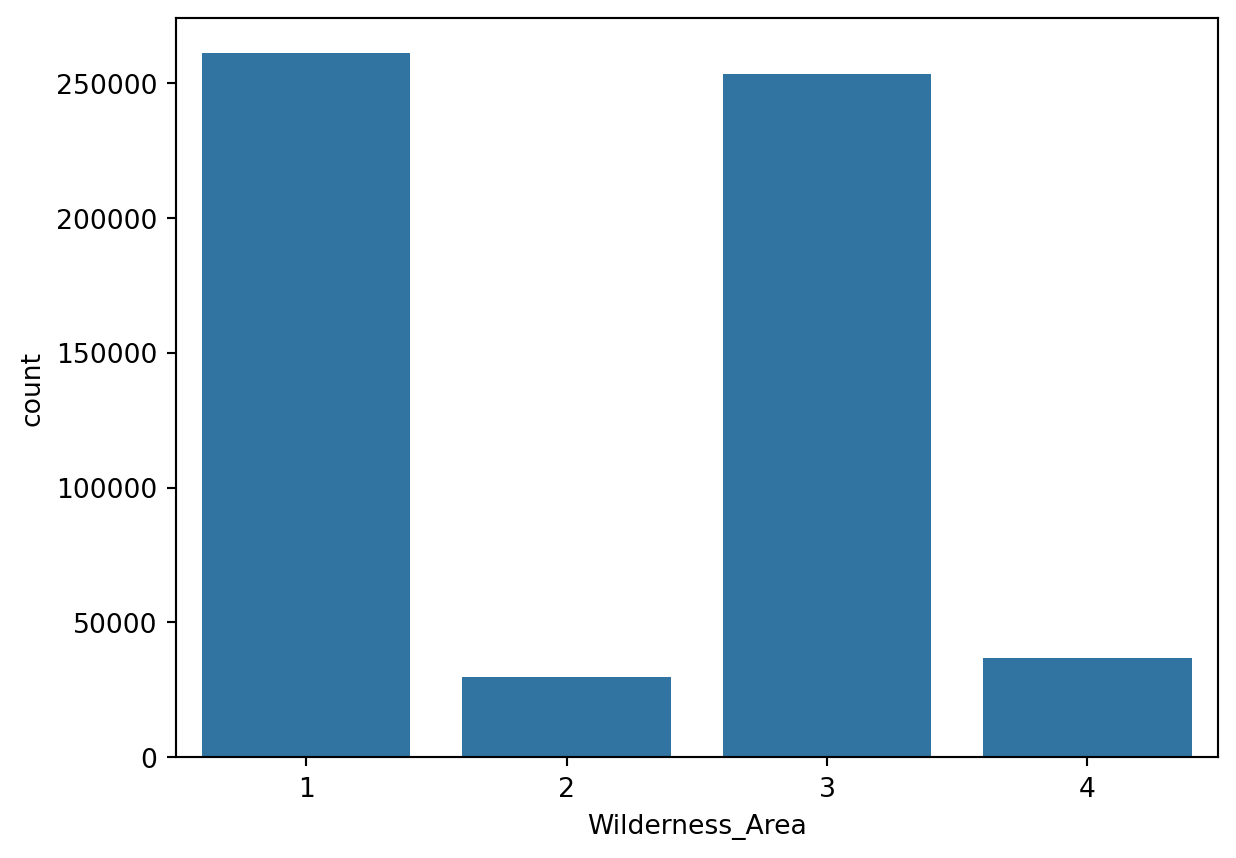
On peut voir que la variable est catégorielle. Elle contient 4 classes équilibrées. Passons à la deuxième variable.
sns.countplot(x='Soil_Type', data=Dataset2)
plt.show()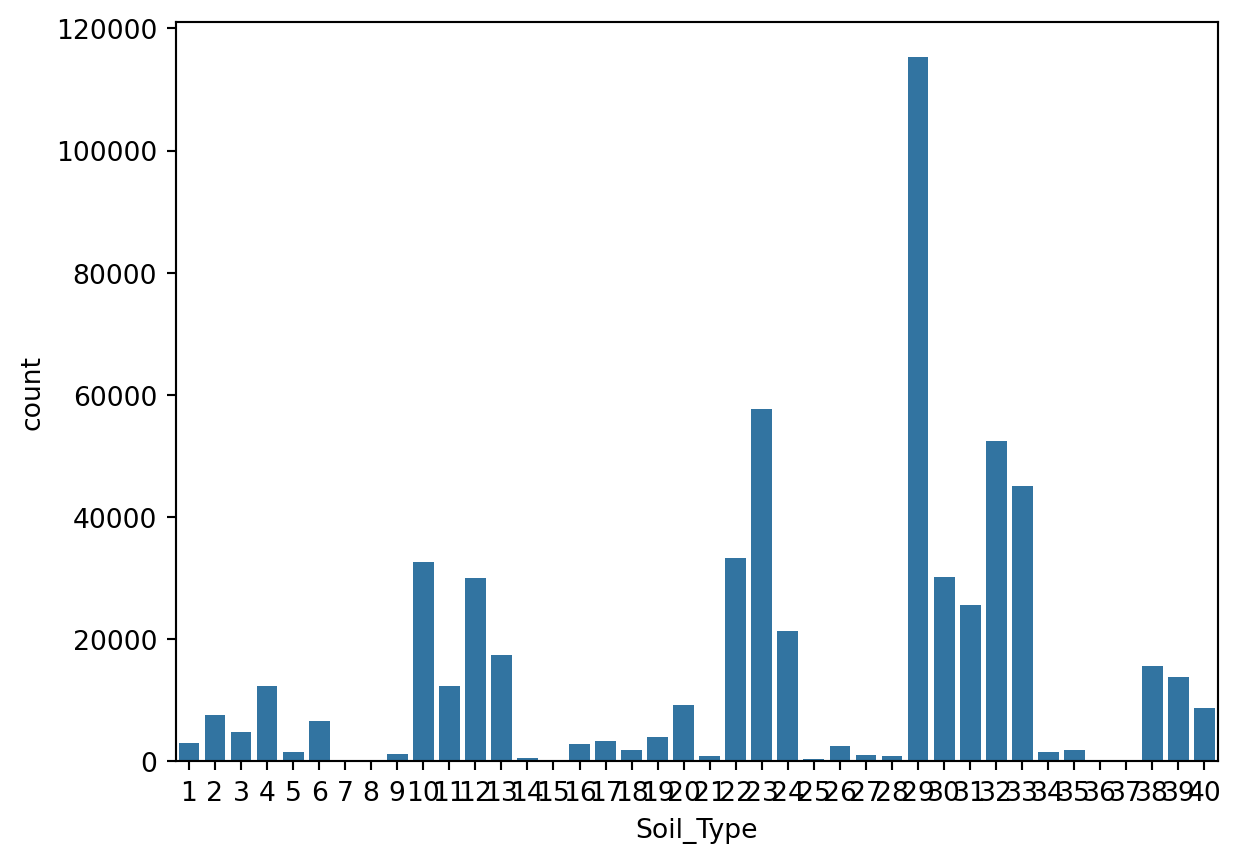
Ici, nous pouvons voir que la variable est plutôt numérique. Traçons la distribution de la variable.
_=Dataset2[['Soil_Type']].hist(figsize=(20, 14))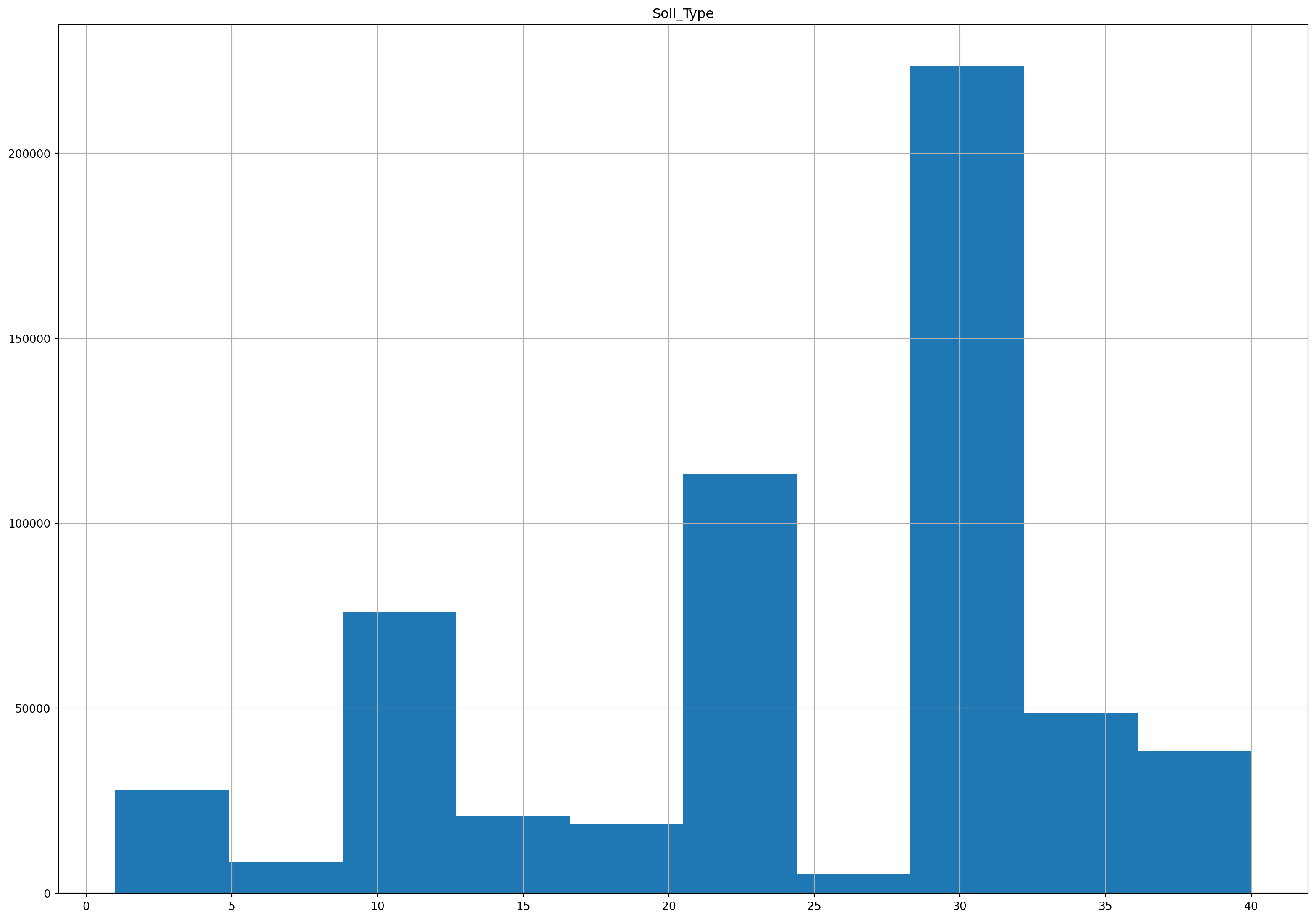
Comme les deux base de données,on les mêmes lignes, nous allons les concaténer.
combined_X = pd.concat([X, Dataset2], axis=1)
combined_X.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 581012 entries, 0 to 581011
Data columns (total 12 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Slope 581012 non-null int64
1 Hillshade_9am 581012 non-null int64
2 Hillshade_Noon 581012 non-null int64
3 Horizontal_Distance_To_Roadways 581012 non-null int64
4 Hillshade_3pm 581012 non-null int64
5 Vertical_Distance_To_Hydrology 581012 non-null int64
6 Horizontal_Distance_To_Fire_Points 581012 non-null int64
7 Aspect 581012 non-null int64
8 Horizontal_Distance_To_Hydrology 581012 non-null int64
9 Elevation 581012 non-null int64
10 Wilderness_Area 581012 non-null int64
11 Soil_Type 581012 non-null int64
dtypes: int64(12)
memory usage: 53.2 MBNous pouvons maintenant séparer les données en train et test. Avec stratification sur la variable cible.
# split data into training and testing sets
print("Splitting data into training and testing sets...")
X_train, X_test, Y_train, Y_test = train_test_split(combined_X, Y, test_size=0.2, random_state=42, stratify=Y)Splitting data into training and testing sets...Avant de passer à la modélisation, nous allons standardiser les données, et encoder les variables catégorielles.
D’abord, nous allons séparer les variables numériques et catégorielles.
X_train.columnsIndex(['Slope', 'Hillshade_9am', 'Hillshade_Noon',
'Horizontal_Distance_To_Roadways', 'Hillshade_3pm',
'Vertical_Distance_To_Hydrology', 'Horizontal_Distance_To_Fire_Points',
'Aspect', 'Horizontal_Distance_To_Hydrology', 'Elevation',
'Wilderness_Area', 'Soil_Type'],
dtype='object')categorical_cols = ['Wilderness_Area']
numerical_cols = ['Slope', 'Hillshade_9am', 'Hillshade_Noon',
'Horizontal_Distance_To_Roadways', 'Hillshade_3pm',
'Vertical_Distance_To_Hydrology', 'Horizontal_Distance_To_Fire_Points',
'Aspect', 'Horizontal_Distance_To_Hydrology', 'Elevation', 'Soil_Type']Nous allons créer un pipeline pour standardiser les données numériques et encoder les variables catégorielles. J’adore les pipelines.
numerical_transformer = Pipeline(steps=[
('imputer', SimpleImputer(strategy='mean')), # or median
('scaler', StandardScaler())])
categorical_transformer = Pipeline(steps=[
('imputer', SimpleImputer(strategy='most_frequent')),
('onehot', OneHotEncoder(handle_unknown='ignore'))])
preprocessor = ColumnTransformer(
transformers=[
('num', numerical_transformer, numerical_cols),
('cat', categorical_transformer, categorical_cols)])
X_train_transformed = preprocessor.fit_transform(X_train)
X_test_transformed = preprocessor.transform(X_test)Ici nous n’avons pas de valeurs manquantes, mais nous avons quand même utilisé un imputer pour remplacer les valeurs manquantes par la moyenne pour les variables numériques et par la valeur la plus fréquente pour les variables catégorielles. Un premier pipeline pour les variables numériques et un deuxième pour les variables catégorielles. Nous avons utilisé un onehot encoder pour les variables catégorielles. Les données sont maintenant prêtes pour la modélisation.
Modélisation
Elastic Net Regression
C’est une méthode de machine learning qui combine la régression Ridge et Lasso. Elle est utilisée pour résoudre le problème de surraprentissage, la multicollinéarité et la sélection de variables.
Passons à sa modélisation :
Nous avons utilisé ici un modèle de régression logistique avec une pénalité elasticnet. Nous avons utilisé une validation croisée pour trouver le meilleur paramètre de régularisation. Nous avons utilisé une pénalité elasticnet avec un ratio de 0.5. Nous avons utilisé un solver saga qui est adapté aux problèmes multiclasse. Nous avons utilisé une tolérance de 0.01. Nous avons utilisé un random state de 12345.
clf_l1l2_LR = LogisticRegressionCV(penalty='elasticnet', l1_ratios=[0.5],
cv=5, multi_class="multinomial",
solver="saga",tol=0.01, random_state=12345)
model = Pipeline(steps=[('preprocessor', preprocessor), ('logistic', clf_l1l2_LR)])
model.fit(X_train,Y_train)
prediction = model.predict(X_test)
accuracy_LR = accuracy_score(Y_test, prediction)
print("Accuracy of Logistic Regression :","%.3f" % accuracy_LR)Accuracy of Logistic Regression : 0.714J’ai un accuracy de 0.714. Ce n’est pas mal. Nous pouvons voir la matrice de confusion.
# Confusion matrix
# Compute the confusion matrix
conf_matrix = confusion_matrix(Y_test, prediction)
# Display the confusion matrix using Seaborn's heatmap
plt.figure(figsize=(10, 7))
sns.heatmap(conf_matrix, annot=True, fmt='g', cmap='Blues')
plt.title('Confusion Matrix')
plt.xlabel('Predicted labels')
plt.ylabel('True labels')
plt.show()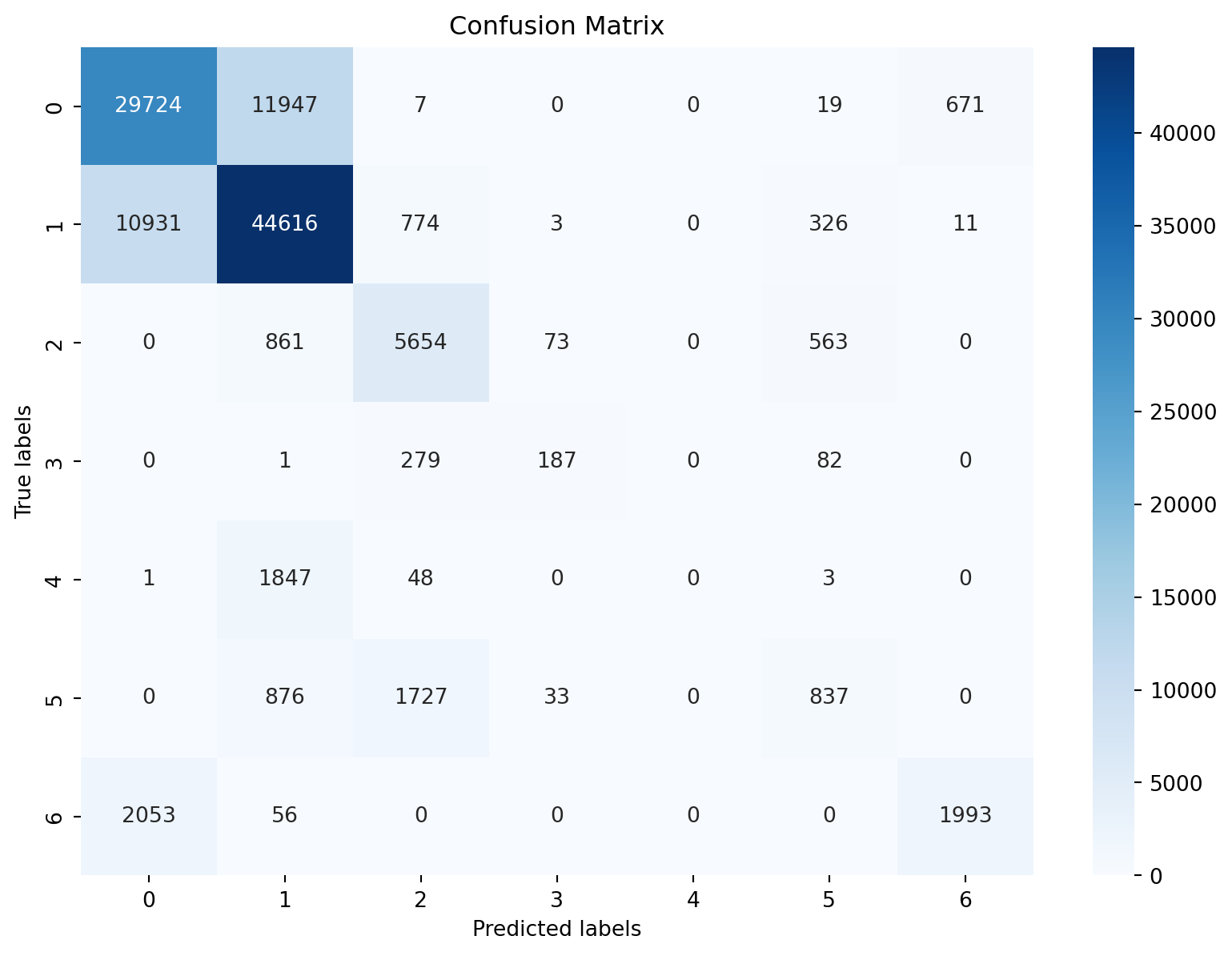
- Les valeurs sur la diagonale principale (de haut à gauche à bas à droite) représentent le nombre de prédictions correctes pour chaque classe. Par exemple, il y a 29724 prédictions correctes pour la classe 0, 44616 pour la classe 1, et ainsi de suite.
- Les valeurs hors de la diagonale indiquent les erreurs de classification. Par exemple, 11947 instances de la classe 0 ont été incorrectement prédites comme appartenant à la classe 1.
- La classe 0 a le plus grand nombre de faux positifs, c’est-à-dire que de nombreuses instances d’autres classes ont été incorrectement prédites comme appartenant à la classe 0.
- Les classes avec le moins de prédictions incorrectes (et donc les plus sombres dans la visualisation) sont la classe 3 et la classe 6, avec respectivement 187 et 1993 prédictions correctes. Les cases avec un fond plus clair, en dehors de la diagonale, indiquent des erreurs moins fréquentes entre les classes spécifiques.
Random Forest
OOB error (Out-of-bag error)
OOB est une méthode de validation croisée pour les forêts aléatoires. Chaque arbre dans la forêt est construit à partir d’un échantillon bootstrap du jeu de données d’entraînement. Certaines observations sont laissées de côté et non utilisées dans la construction d’un arbre donné. Ces observations “hors sac” peuvent être utilisées pour évaluer les performances de cet arbre. Du coup on peut utiliser cette méthode pour évaluer la performance de la forêt aléatoire et ajuster les hyperparamètres.
Le code ci-dessous montre comment calculer l’erreur OOB pour un modèle de forêt aléatoire. Il permet en particulier de sélectionner la profondeur de l’arbre dans la forêt aléatoire. Le modèle de forêt aléatoire est entraîné avec une profondeur d’arbre de 10, 20 et 30. L’erreur OOB est calculée pour chaque modèle. Le modèle avec la plus petite erreur OOB est sélectionné.
## Training Random Forest
depths = [10, 20, 30]
oob_errors = []
models = []
best_oob_error = float('inf')
best_model = None
i = 0
for depth in depths:
print(i)
model = RandomForestClassifier(max_depth=depth, oob_score=True, random_state=42,
n_estimators=100,
warm_start=True # This allows us to add more estimators later if needed
)
model.fit(X_train, Y_train)
oob_error = 1 - model.oob_score_
oob_errors.append(oob_error)
models.append(model)
if oob_error < best_oob_error:
best_oob_error = oob_error
best_model = model
i = i+1
print("Done")
# Print OOB errors for each model
for depth, error in zip(depths, oob_errors):
print(f"Depth: {depth}, OOB Error: {error}")0
Done
1
Done
2
Done
Depth: 10, OOB Error: 0.21680518234371537
Depth: 20, OOB Error: 0.05706860237215716
Depth: 30, OOB Error: 0.038628770096964526Accuracy et matrice de confusion
# Compute the accuracy of the best random forest model
predictions = best_model.predict(X_test)
accuracy = accuracy_score(Y_test, predictions)
print("Accuracy of the Best Random Forest: {:.3f}".format(accuracy))
# Display the confusion matrix
conf_matrix = confusion_matrix(Y_test, predictions)
plt.figure(figsize=(10, 7))
sns.heatmap(conf_matrix, annot=True, fmt='g', cmap='Blues')
plt.title('Confusion Matrix')
plt.xlabel('Predicted labels')
plt.ylabel('True labels')
plt.show()Accuracy of the Best Random Forest: 0.962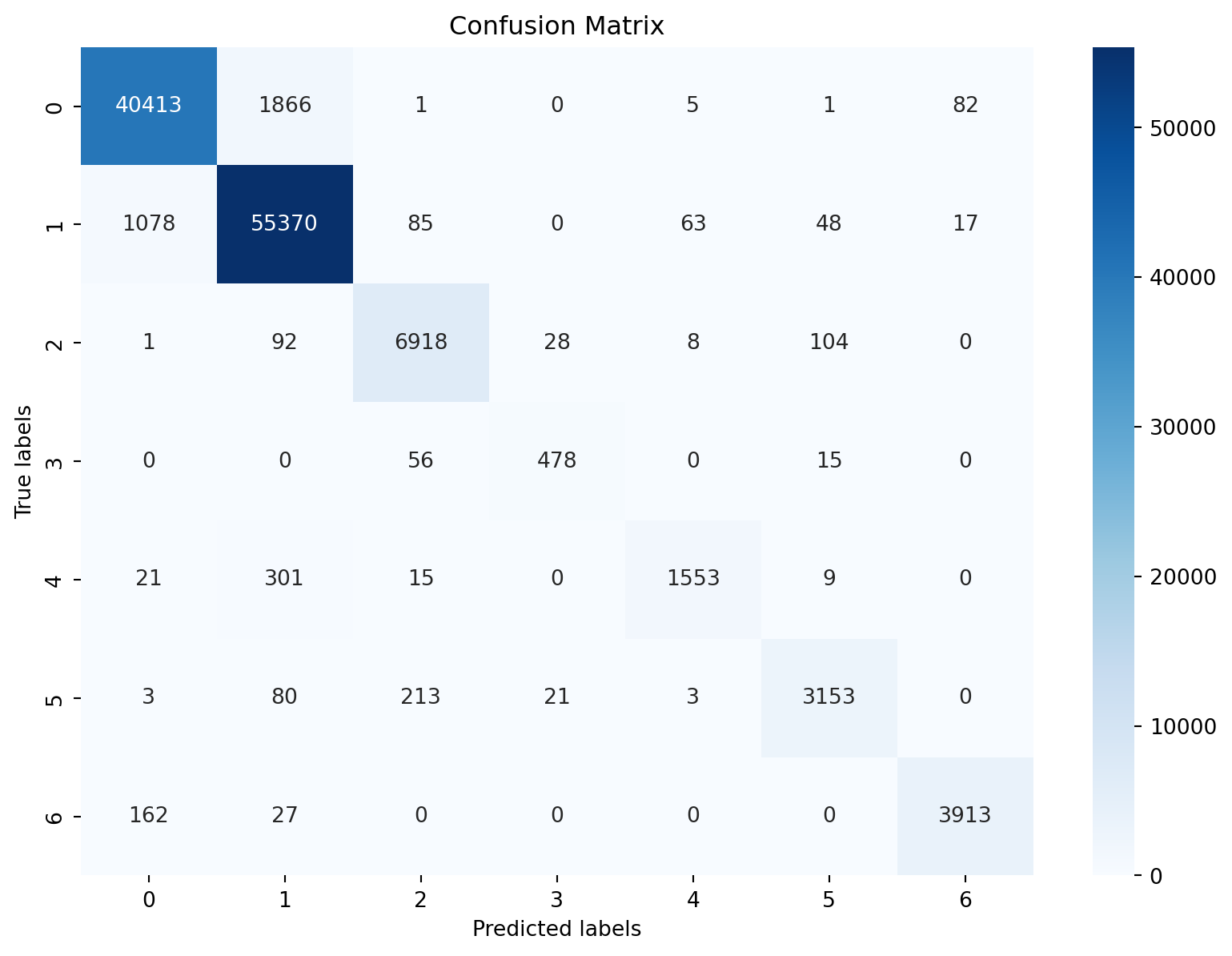
Nous avons un accuracy de 0.962. C’est très bien si on compare avec le modèle de régression logistique qui a un accuracy de 0.714. Nous pouvons voir la matrice de confusion.
Xgboost
Essayons tout d’abord par expliquer ce qu’est le Xgboost. Xgboost signifie Extreme Gradient Boosting. Il combine des weaks models afin de produire des prédictions plus précises. Il est très rapide et performant.
Avant de passer à la modélisation, il faut transformer les données en un format spécifique à Xgboost. En effet, le package xgboost ne gère pas les chaînes de caractères pour les étiquettes contrairement à tous les modèles entraînés précédemment, donc vous devez d’abord les encoder en tant qu’entiers. De plus, il encode automatiquement les étiquettes en tant que 0, 1, 2, etc. Cela signifie que si vous avez des étiquettes de classe 1, 2, 3, 4, 5, 6, 7, vous devez les encoder en tant que 0, 1, 2, 3, 4, 5, 6. Il faut le faire pour les labels(Y_train et Y_test) d’entraînement et de test.
# encode string class values as integers
label_encoder = LabelEncoder()
label_encoder = label_encoder.fit(Y)
label_encoded_y_train = label_encoder.transform(Y_train)
label_encoded_y_test = label_encoder.transform(Y_test)# Vérifiez les étiquettes uniques dans les ensembles d'entraînement et de test
unique_train_labels = np.unique(label_encoded_y_train)
unique_test_labels = np.unique(label_encoded_y_test)
print("Unique labels in training set:", unique_train_labels)
print("Unique labels in test set:", unique_test_labels)Unique labels in training set: [0 1 2 3 4 5 6]
Unique labels in test set: [0 1 2 3 4 5 6]Nous avons utilisé un modèle de classification Xgboost avec les paramètres suivants:
objective=‘multi:softprob’ : Spécifie la fonction objectif de l’entraînement. Ici, ‘multi:softprob’ est utilisé pour les problèmes de classification multiclasse et retournera une matrice de probabilité estimée pour chaque classe, ce qui est nécessaire pour calculer des scores comme le log-loss.
seed=‘12345’ : Fournit une graine pour le générateur de nombres aléatoires. Cela garantit que les résultats sont reproductibles. Toutefois, il semble y avoir une petite confusion ici, car la graine devrait être un entier (seed=12345), pas une chaîne de caractères (seed=‘12345’).
gamma=0 : Paramètre de régularisation qui minimise la complexité du modèle et aide à prévenir le surajustement. La valeur de 0 indique qu’il n’y a pas de régularisation supplémentaire.
learning_rate=0.05 : C’est le taux d’apprentissage, également connu sous le nom d’eta. Cela contribue à rendre le processus d’apprentissage plus robuste en empêchant les poids de s’ajuster trop fortement à chaque itération. Une valeur plus faible peut nécessiter plus d’arbres pour apprendre les mêmes relations, mais peut améliorer la performance finale du modèle.
max_depth=5 : Détermine la profondeur maximale de chaque arbre. C’est un autre paramètre qui aide à prévenir le surajustement. Plus la profondeur est grande, plus le modèle est complexe.
n_estimators=200 : Le nombre d’arbres à construire. Plus il y a d’arbres, plus le modèle peut être précis, mais cela augmente aussi le temps de calcul et le risque de surajustement.
clf_xgb = XGBClassifier(objective='multi:softprob', seed='12345',
gamma=0, learning_rate=0.05, max_depth=5, n_estimators=200,num_class=7)
clf_xgb.fit(X_train, label_encoded_y_train)
accuracy_xgb = accuracy_score(label_encoder.transform(Y_test), clf_xgb.predict(X_test))
print("Accuracy of XGBOOST :","%.3f" % accuracy_xgb)Accuracy of XGBOOST : 0.786Ici on a un accuracy de 0.0.786. C’est mieux que le modèle de régression logistique mais moins bien que le modèle de forêt aléatoire. Nous pouvons voir la matrice de confusion.
# Display the confusion matrix
predictions = clf_xgb.predict(X_test)
conf_matrix = confusion_matrix(label_encoded_y_test, predictions)
plt.figure(figsize=(10, 7))
sns.heatmap(conf_matrix, annot=True, fmt='g', cmap='Blues')
plt.title('Confusion Matrix')
plt.xlabel('Predicted labels')
plt.ylabel('True labels')
plt.show()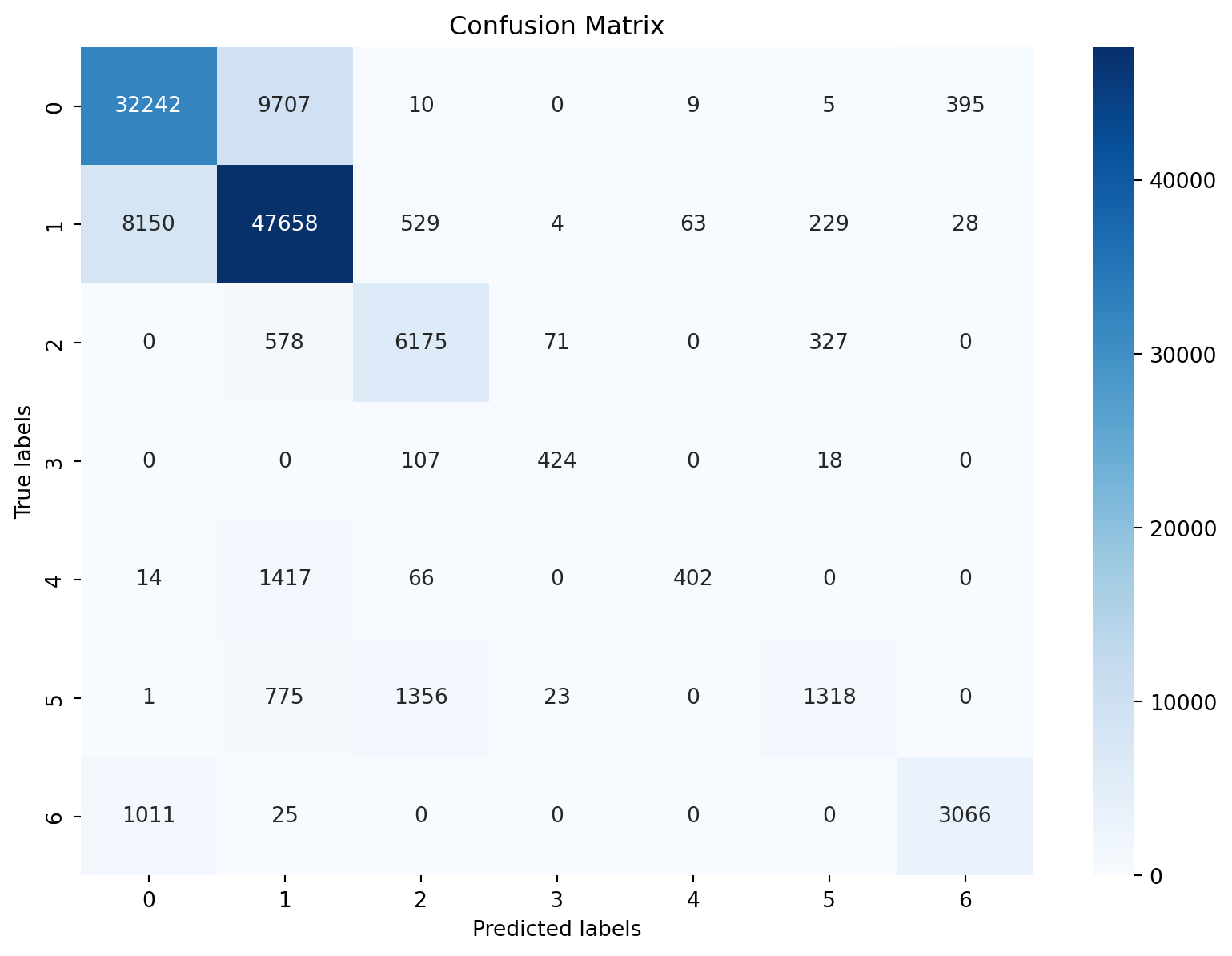
Courbe ROC
La courbe ROC est un graphique qui montre la performance d’un modèle de classification à différents seuils de classification. Elle trace le taux de vrais positifs (TPR) en fonction du taux de faux positifs (FPR) à différents seuils de classification. Le TPR est également connu sous le nom de rappel et le FPR est égal à 1 - spécificité.
import numpy as np
from sklearn.preprocessing import label_binarize
from sklearn.metrics import roc_curve, auc
def compute_roc_auc(models, X, Y_test):
"""
Compute ROC AUC for a list of models.
Args:
models (dict): A dictionary of models with their names as keys.
X_test (array-like): Test features.
Y_test (array-like): True labels for the test set.
Returns:
dict: A dictionary containing FPR, TPR, and ROC AUC for each model.
"""
Y_classes = np.unique(Y_test)
Y_test_binarized = label_binarize(Y_test, classes=Y_classes)
n_classes = len(Y_classes)
results = {}
for model_name, model in models.items():
if model_name == 'Logistic':
model = Pipeline(steps=[('preprocessor', preprocessor), ('logistic', clf_l1l2_LR)])
model.fit(X_train,Y_train)
score = model.predict_proba(X_test)
else:
score = model.predict_proba(X_test)
fpr = dict()
tpr = dict()
roc_auc = dict()
# Compute ROC for each class
for i in range(n_classes):
fpr[i], tpr[i], _ = roc_curve(Y_test_binarized[:, i], score[:, i])
roc_auc[i] = auc(fpr[i], tpr[i])
# Compute micro-average ROC curve and ROC area
fpr["micro"], tpr["micro"], _ = roc_curve(Y_test_binarized.ravel(), score.ravel())
roc_auc["micro"] = auc(fpr["micro"], tpr["micro"])
results[model_name] = {'fpr': fpr, 'tpr': tpr, 'roc_auc': roc_auc, 'score':score}
return results
models = {
'Logistic': clf_l1l2_LR,
# Assurez-vous que clf_svm est entraîné sur des données mises à l'échelle si nécessaire
'Random Forests': best_model,
'XGBOOST': clf_xgb,
}
roc_results = compute_roc_auc(models, X_test, Y_test)import matplotlib.pyplot as plt
plt.figure(figsize=(6, 6))
plt.plot([0, 1], [0, 1], 'k--') # Ligne diagonale
for model_name, metrics in roc_results.items():
plt.plot(metrics['fpr']['micro'], metrics['tpr']['micro'], label=f'{model_name} Micro (area = {metrics["roc_auc"]["micro"]:.2f})')
plt.legend()
plt.xlabel("False Positive Rate")
plt.ylabel("True Positive Rate")
plt.title('Micro-Average Receiver Operating Characteristic')
plt.show()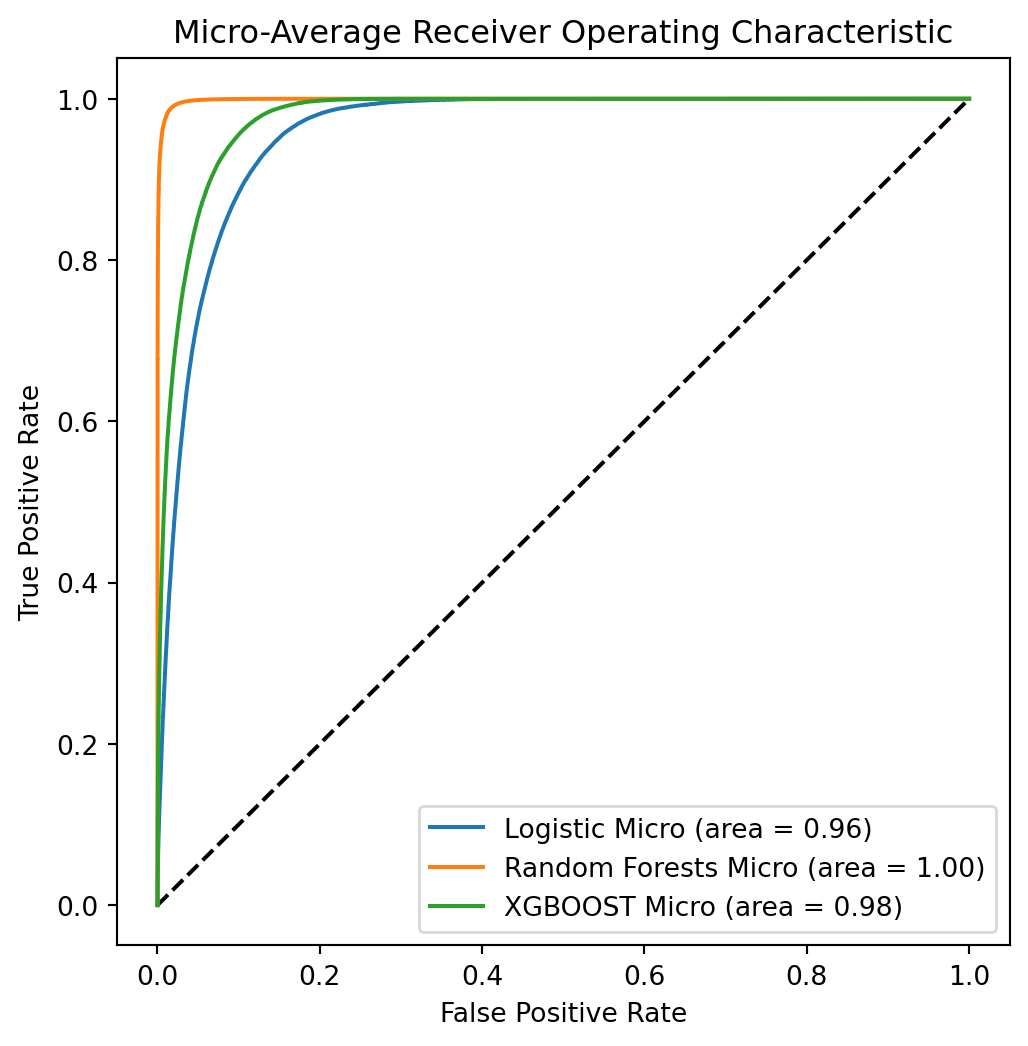
La courbe ROC montre que le modèle de forêt aléatoire est le meilleur modèle. Il a la plus grande surface sous la courbe (AUC). Le modèle Xgboost est le deuxième meilleur modèle. Le modèle de régression logistique est le moins bon modèle.
Bonus
from sklearn.metrics import roc_auc_score
# Affichage des AUC micro-averaged
print("Area under Roc Curve (micro-average) for:\n")
for model_name in models.keys():
print(f"- {model_name}: {roc_results[model_name]['roc_auc']['micro']:.3f}")
# Calcul et affichage des AUC one-vs-one macro-averaged
print("\nArea under Roc Curve (one-vs-one macro-average) for:\n")
for model_name, model in models.items():
score = roc_results[model_name]['score']
auc_ovo_macro = roc_auc_score(Y_test, score, multi_class="ovo", average="macro")
print(f"- {model_name}: {auc_ovo_macro:.3f}")Area under Roc Curve (micro-average) for:
- Logistic: 0.959
- Random Forests: 0.999
- XGBOOST: 0.977
Area under Roc Curve (one-vs-one macro-average) for:
- Logistic: 0.917
- Random Forests: 0.998
- XGBOOST: 0.966Conclusion
Pour aborder cet exercice de manière efficace, je vous recommande fortement de vous concentrer sur la préparation préalable de vos propres fonctions, plutôt que de vous reposer excessivement sur des générateurs de texte automatiques. La création de vos fonctions en amont vous permettra de gagner un temps précieux. N’oubliez pas que les fonctions et les pipelines que vous avez déjà utilisés ont été préparés avant même de commencer le devoir ; votre tâche consiste essentiellement à les adapter aux besoins spécifiques de la situation.
Par ailleurs, l’utilisation judicieuse de ChatGPT peut s’avérer très bénéfique, notamment dans des tâches telles que la génération de boxplots ou l’implémentation de l’algorithme pour calculer l’erreur out-of-bag (Out-of-bag error). ChatGPT peut fournir des conseils, des exemples de code ou des explications qui peuvent faciliter le processus de développement. Cependant, gardez à l’esprit que l’outil doit être utilisé comme un complément à votre propre travail de programmation et de réflexion, et non comme une solution complète.